독성 예측(Toxicity Prediction) 연구는 약물 개발 및 화학 물질의 안정성 평가에서 중요한 역할을 하며 기계 학습 기반의 접근 방식은 지속적으로 연구되어 왔다 (Cavasotto et al., 2022). 초기 독성 예측 연구에서는 일반적으로 화학 물질의 분자 구조를 이진 벡터로 표현한 분자 지문(Molecular Fingerprints)를 사용하여 기계 학습 모델에 입력 데이터로 사용하였다 (Pu et al., 2019). 이후, 다양한 딥러닝(Deep Learning) 연구가 발전되어 옴에 따라 단순히 이진 벡터로 변환하는 방식이 아닌 분자를 Graph로 간주하여 표현하는 연구가 진행되고 있다 (Guo et al., 2023). 분자는 원자와 원자간의 연결로 이루어지는데, 원자를 Graph의 Node, 원자간의 연결을 Edge로 간주한다. 분자를 Graph로 나타낸 예시는 다음과 같다.
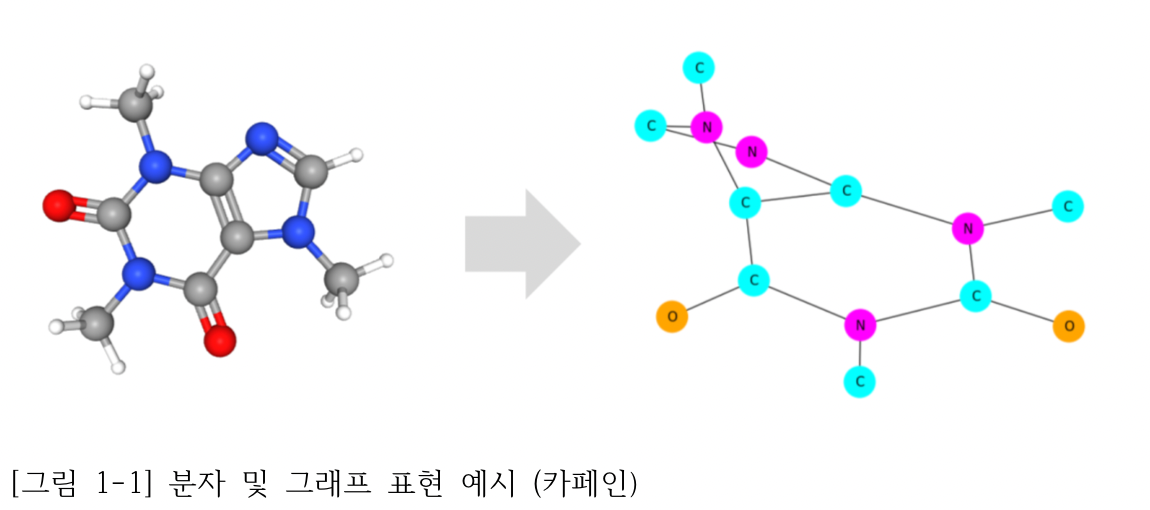
기존 방식과 비교하였을 때, 해당 방식은 분자의 구조적 정보를 잘 반영할 수 있다는 장점이 있다. 분자의 구조적 정보란 분자의 원자 배열, 결합 형식, 분자 모양, 분자 크기 등을 포함하는데, 분자의 독성은 이 구조적 특성에 따라 달라질 수 있다. 예를 들어, 분자가 특정 생체 분자와 결합할 수 있는 구조를 가지고 있다면 독성을 가지기 쉬워진다. 따라서 분자의 구조적 정보를 잘 파악하는 것이 매우 중요하다 (Wei et al., 2021).
분자 그래프 데이터는 Graph Neural Network(GNN) 모델을 활용하여 그 정보를 추출할 수 있다 (Scarselli et al., 2008). 초기의 그래프 기반 모델은 상대적으로 단순한 그래프 구조에 집중했지만, 최근 연구는 더 복잡한 그래프 표현과 학습 방식에 대한 연구가 진행되고 있다. GNN 계열 모델은 크게 Graph Convolution Network(GCN)과 Graph Isomorphism Network(GIN)으로 나눌 수 있다. GCN은 Convolution 신경망(CNN)의 개념을 그래프 구조 데이터에 적용한 것으로, 그래프의 노드에 히든 벡터를 할당하고, 히든 벡터를 Graph Convolution 연산을 통해 업데이트 한다 (Kipf et al., 2016). GIN은 그래프 순환을 기반으로 하는 모델이다. 그래프 순환은 Recurrent Neural Network(RNN)의 개념을 그래프 구조 데이터에 적용한 것이다. GIN은 그래프 순환을 사용하여 그래프의 노드 정보를 순차적으로 업데이트 하는 모델로 자기 동형성을 고려한다. 자기 동형성은 두 그래프가 동일한 구조를 가지고 있는 경우를 의미하며 이를 활용하여 그래프의 특성을 보다 잘 수집할 수 있다 (Xu et al., 2018).
그러나 분자 독성 예측에서 GNN 계열 모델을 적용하려할 때, 두 가지 어려움이 존재한다. 첫 번째는 지도 학습을 위한 독성 여부(독성 있음/ 없음) 레이블이 부족한 분자가 많다는 점이다. 그 이유는 분자의 독성 여부를 실험을 통해 밝혀내고 데이터셋을 레이블링하기 위해서는 많은 인적, 시간적, 비용적 자본이 소요되기 때문이다. 두 번째는 기존의 분자 데이터로 학습된 GNN 모델은 새롭게 합성된 분자에 대해서는 일반화 능력이 부족할 수 있기 때문이다. 분자의 구조가 복잡하기 때문에 단순 GNN 구조로는 정보를 인코딩하기 쉽지 않고, 이에 따라 일반화 능력이 부족할 수 밖에 없다.
따라서 해당 한계점을 극복하기 위해 GROVER(Graph Representation frOm self-superVised mEssage passing tRansformer) 모델이 제안되었다. 해당 모델은 자기 지도 학습을 통해 레이블이 없는 수많은 분자 데이터로부터 분자의 구조적, 의미적 정보를 학습하였으며, Message Passing Networks를 Transformer 구조와 결합하여 분자의 표현력을 높일 수 있는 인코더를 구성하였다 (Rong et al., 2020). 우리는 기존 GNN 모델을 보완한 GROVER 모델을 사용하여 분자 정보를 추출하고자 한다.
분자 정보 추출을 위해, 그래프 기반 피쳐 추출법 외에 텍스트 기반 접근법 또한 연구되고 있다. 처음 그래프 기반 접근법이 제안된 이후, 독성 예측을 포함한 많은 분야에서 더 나은 성능을 보임이 확인되었지만 실험을 통해 검증된 대규모의 레이블 분자 데이터셋을 구축해야한다는 단점을 가지고 있다 (Duvenaud et al., 2015). 따라서, 소규모 데이터셋만으로도 좋은 성능을 구현할 수 있는 SMILES Transformer가 제안되었다. 해당 모델은 분자를 표현하는 문자열인 SMILES식을 활용한 텍스트 기반 접근법을 활용하여 분자의 특성을 소규모 데이터셋만으로도 잘 추출할 수 있도록 하였다 (Honda et al., 2019). 따라서 텍스트 기반 피쳐 추출법으로 SMILES Transformer 모델을 선정하여 분자 정보를 추출하고자 한다.
'AI > Toxicity Prediction' 카테고리의 다른 글
| [Toxicity Prediction] 분자를 graph로 표현하기/ graph representation (1) | 2023.05.09 |
|---|---|
| [Toxicity Prediction] Class imbalance와 SMOTE (0) | 2023.05.05 |
| [Toxicity Prediction] fingerprint / ECEP(extended-connectivity fingerprint) (0) | 2022.06.06 |
| [Toxicity Prediction] 파이썬으로 SDF 파일 읽기/ 분자 데이터 SMILES식 확인하기 (0) | 2022.05.02 |
| [Toxicity Prediction]Graph Learning / molecule (분자) data training의 과제 (0) | 2022.04.17 |
